反復測定分散分析:repeated measures ANOVA (混合効果モデル)
参考文献(折笠 2016)の図10の結果。
ランダム切片モデル(各 ID ごとのランダム切片を線形回帰に取り入れたモデル)。これにより、ベースラインにおける個々の被験者の違い(ランダム効果)を調整 しつつ、固定効果である sequence, treatment, period の影響を適切に分析できます。
fit3 <- lmer(
point ~ sequence + treatment + period + (1|ID),
data = data
)
anova(fit3)
他のモデルと比較
分散分析表 (Between Subject) の平方和。Between SubjectですのでIDを説明変数とした線形回帰モデル。ID を固定効果として扱うことで、各被験者ごとの違いをモデルに組み込む。ただし、被験者ごとの変動を推定するのではなく、ダミー変数として扱うため、データ数が増えるとパラメータ数も増え、モデルが過学習しやすくなる。
fit4 <- lm(
point ~ sequence + ID,
data = data
)
anova(fit4)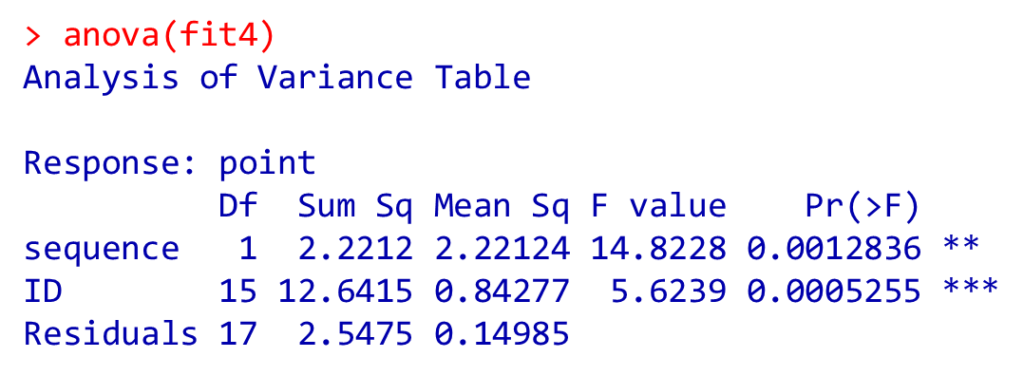
分散分析表 (Within-subject) の平方和。Within-subjectですのでIDを変量効果とした混合効果モデル。ベースラインにおける個々の被験者の違い(ランダム効果)を調整 しつつ、treatment と period の影響を評価する。
fit5 <- lmer(
point ~ treatment + period + (1|ID),
data = data
)
anova(fit5)
比較
anova(fit3, fit4)
fit3 の方が明らかに良い(p < 0.001)。 ID はランダム効果で扱うべき。
anova(fit3, fit5)
anova(fit3, fit5) では sequence の影響が弱め(p = 0.097)だが、完全に無視できるとは言えない。
事後検定
参考文献(折笠 2016)の図11の結果。分散分析を適用します。
$\blacklozenge$ 効果の検定
fit6 <- lm(
point ~ period + treatment + ID,
data = data
)
anova(fit6)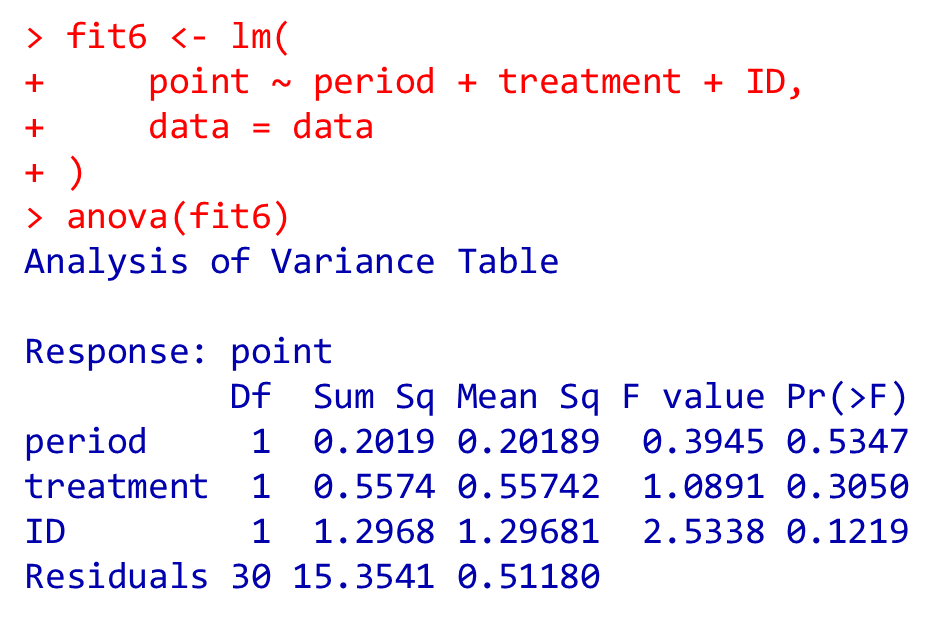

periodの結果が参考文献(折笠 2016)の図11の結果と異なります。原因が分かり次第修正します。
最小二乗平均
lmerTestのls_means関数を使用
ls_means(fit5)
$\blacklozenge$ I期、II期、治療A、治療Bの平均
mean(data[data$period=="I", "point"])
mean(data[data$period=="II", "point"])
mean(data[data$treatment=="A", "point"])
mean(data[data$treatment=="B", "point"])
最小二乗平均差
lmerTestのdifflsmeans関数。参考文献に基準を合わせます。
data$period <- relevel(data$period, "I")
data$treatment <- relevel(data$treatment, "A")
levels(data$period)
levels(data$treatment)
fit5_2 <- lmer(
point ~ treatment + period + (1|ID),
data = data
)
difflsmeans(fit5_2)
求め方・・・
$\blacklozenge$ 各群の最小二乗法推定量
繰り返しありと仮定して二元配置分散分析から求めます
data$period <- relevel(data$period, "II")
data$treatment <- relevel(data$treatment, "B")
levels(data$period)
levels(data$treatment)
fit7 <- lm(
point ~ period + treatment,
data=data
)
summary(fit7)$coef
Y11(IのA):1.9611029 + 0.1390278 – 0.2565278 = 1.843603
Y12(IIのB):1.9611029
Y21(IのB):1.9611029 + 0.1390278 = 2.100131
Y22(IIのA):1.9611029 – 0.2565278 = 1.704575
I期:(1.8436029+2.100131)/2=1.971867
II期:(1.961103+1.704575)/2=1.832839
治療A:(1.8436029+1.704575)/2=1.774089
治療B:(1.961103+2.100131)/2=2.030617
差の平均の最小二乗法推定量
I期-II期:0.1390278
治療A-治療B:-0.2565278
差の平均の最小二乗法推定量の95%信頼区間
confint ( fit7 , level = 0.95 )
参考文献
折笠秀樹. クロスオーバー試験の計画および解析. 薬理と治療, 2016, 44.9: 1261-1276.
角間辰之, 服部聡; 臨床試験のデザインと解析: 薬剤開発のためのバイオ統計. 近代科学社. 2012

