分割表の基礎
分割表を作成します。
results = 検査結果(positive = 陽性、negative=陰性)
affected = 罹患(0=無し、1=あり)
results <- c(rep("negative",70), rep("positive",75))
affected <- c(
rep("no", 45),
rep("yes", 25),
rep("yes", 8),
rep("no", 67)
)
tab <- xtabs(~ results + affected)
print(tab)> print(tab)
affected
results no yes
negative 45 25
positive 67 8レベルの確認、基準の変更
factor() 関数は名義変数をカテゴリカル変数(因子型変数)に変換し、データのカテゴリーは「水準」として定義されます。levels() 関数で表示されるカテゴリカル変数の水準のうち、最初に表示されるカテゴリーがデフォルトで基準カテゴリー(参照カテゴリー)となり、統計モデルでの比較の基点として機能します。relevel() 関数や factor() 関数の levels 引数を使用することで、レベルの順序を指定し、基準カテゴリーを変更することが可能です。
results <- factor(results)
affected <- factor(affected)
levels(results)
levels(affected)> results <- factor(results)
> affected <- factor(affected)
> levels(results)
[1] "negative" "positive"
> levels(affected)
[1] "no" "yes"先頭に記載されている方がデータの基準カテゴリーとなります。検査結果の基準は”negative”、罹患の基準は”no”になっています。基準カテゴリーを変更して、左上が検査結果「陽性」、罹患「あり」に配置されるように変更します。
results <- relevel(results, "positive")
affected <- relevel(affected , "yes")
levels(results)
levels(affected)> levels(results)
[1] "positive" "negative"
> levels(affected)
[1] "yes" "no" tab2 <- xtabs(~ results + affected)
print(tab2)> print(tab2)
affected
results yes no
positive 8 67
negative 25 45これで馴染みのある並び方になりました
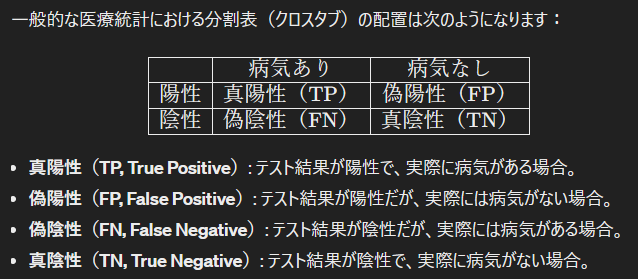
感度=$\dfrac{8}{8+25}=\frac{TP}{TP+FN}$
特異度=$\dfrac{45}{67+45}=\frac{TN}{FP+TN}$
参考(ChatGPTの回答)
分割表を作成
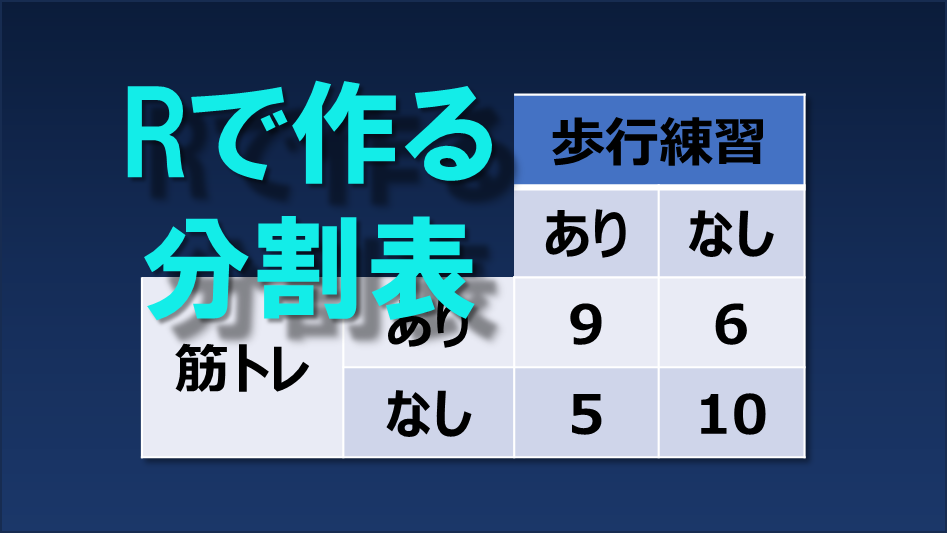
筋力トレーニング:musc(実施=yes、非実施=no)
歩行練習:gait(実施=yes、非実施=no)
効果:effect (あり=good、なし=not)
dat <- data.frame(
c(rep("yes", 15),rep("no", 15)),
c(rep("yes", 9), rep("no", 6), rep("yes", 5), rep("no", 10)),
c(rep("good", 12), rep("not", 3), rep("good", 4), "not", "good", "good", rep("not", 8))
)
colnames(dat) <- c("musc", "gait", "effect")
head(dat)> head(dat)
musc gait effect
1 yes yes good
2 yes yes good
3 yes yes good
4 yes yes good
5 yes yes good
6 yes yes good1列目(筋トレ)と3列目(歩行練習)の分割表を作成
tab1 <- xtabs(
~musc + effect,
data=dat
)
print(tab1)> print(tab1)
effect
musc good not
no 6 9
yes 12 3分割表を作業フォルダに保存する場合
write.csv(tab1, file="tab1.csv",fileEncoding="UTF-8")エクセルなどで開いて文字化けすることが気になるのであれば、以下のように保存
write.csv(tab1, file="tab1.csv",fileEncoding="Shift-JIS")ただし、次回読み込むときも fileEncoding=”Shift-JIS” とする
分割表の操作
周辺合計
addmargins(tab1)> addmargins(tab1)
effect
musc good not Sum
no 6 9 15
yes 12 3 15
Sum 18 12 30基準カテゴリーを変更せずに、行の入れ替えも可能です。
tab1[c("yes","no"), ]> tab1[c("yes","no"), ]
effect
musc good not
yes 12 3
no 6 9転置
t(tab1)> t(tab1)
musc
effect no yes
good 6 12
not 9 3
コメント欄 『間違い』や『分かりにくい部分』などのご意見もお寄せください