基準カテゴリー(参照カテゴリー)
factor() 関数は名義変数をカテゴリカル変数(因子型変数)に変換し、データのカテゴリーは「水準」として定義されます。levels() 関数で表示されるカテゴリカル変数の水準のうち、最初に表示されるカテゴリーがデフォルトで基準カテゴリー(参照カテゴリー)となり、統計モデルでの比較の基点として機能します。relevel() 関数や factor() 関数の levels 引数を使用することで、レベルの順序を指定し、基準カテゴリーを変更することが可能です。
ロジスティック回帰を行うときには、オッズとオッズ比の基準カテゴリーが非常に重要になります。特に日本語記述の場合には、解析を行う前に基準を設定することをお勧めします。
データセットcross-1をdataに格納します(ファイルの読み込み)
xtabs()関数で分割表を作成
data <- read.csv("cross-1.csv", header=T, fileEncoding = "UTF-8")
tab <- xtabs(~ treatment + effect, data = data)
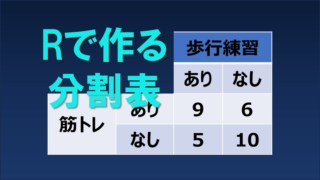
print(tab)> print(tab)
effect
treatment no yes
no 9 6
yes 3 12名義変数(カテゴリカル変数)を factor() 関数を使用して因子型変数に変換。このことで変数に水準(level)が付与されます。何も設定しない場合には、先頭の「no」が基準カテゴリーとして決定されます。
data$treatment <- as.factor(data$treatment)
data$effect <- as.factor(data$effect)
levels(data$treatment)
levels(data$effect)> levels(data$treatment)
[1] "no" "yes"
> levels(data$effect)
[1] "no" "yes"effectを目的変数、treatmentを説明変数としたロジスティック回帰を実行してみましょう。treatment、effectを0, 1の二値変数に変換して(yes=1)、それぞれtreatment_n、effect_nとします。
data$treatment_n <- as.factor(ifelse(data$treatment == "yes", 1 , 0))
data$effect_n <- as.factor(ifelse(data$effect == "yes", 1 , 0))
levels(data$treatment_n)
levels(data$effect_n)0が基準カテゴリーになっています。
> levels(data$treatment_n)
[1] "0" "1"
> levels(data$effect_n)
[1] "0" "1"ロジスティック回帰
effectを目的変数、treatmentを説明変数としたロジスティック回帰の実行。
fit <- glm(formula = effect_n ~ treatment_n, family=binomial, data=data)
summary(fit)> summary(fit)
Call:
glm(formula = effect_n ~ treatment_n, family = binomial, data = data)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -0.4055 0.5270 -0.769 0.4417
treatment_n1 1.7918 0.8333 2.150 0.0315 *
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 40.381 on 29 degrees of freedom
Residual deviance: 35.202 on 28 degrees of freedom
AIC: 39.202
Number of Fisher Scoring iterations: 4オッズ比
exp(1.7918) > exp(1.7918)
[1] 6.000243treatmentの効果は有意なので、treatment=1, つまり「treatmentあり」は「treatmentなし」よりも、$e^{1.7918}$=約6倍のオッズであると言えます。この場合、オッズは、effectの基準カテゴリーが0なので、「effectあり」になる割合ということになります。分割表から求めたオッズは、$\dfrac{(12/3)}{(6/9)}=6$となっています。
目的変数が3値の場合の基準カテゴリー
データセットcross-2をdata2に格納します(ファイルの読み込み)
data2 <- read.csv("cross-2.csv", header=T, fileEncoding = "Shift-JIS")
tab2 <- xtabs(~ treatment + effect, data = data2)
print(tab2)> print(tab2)
effect
treatment no yes
20min/wk 10 2
40min/wk 4 5
60min/wk 1 8レベルの確認
data2$treatment <- as.factor(data2$treatment)
data2$effect <- as.factor(data2$effect)
levels(data2$treatment)
levels(data2$effect)treatmentの基準カテゴリーは「20min/wk」、effectの基準カテゴリーは「no」となっています。
> levels(data2$treatment)
[1] "20min/wk" "40min/wk" "60min/wk"
> levels(data2$effect)
[1] "no" "yes"ここでは、このまま解析を進めてみます。0, 1, 2などに置き換えても、因子型変数であれば結果は同じです。ただし、数値として認識した場合には結果が異なるので注意してください。
fit2 <- glm(effect ~ treatment, family = binomial, data=data2)
summary(fit2)> summary(fit2)
Call:
glm(formula = effect ~ treatment, family = binomial, data = data2)
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -1.6094 0.7746 -2.078 0.03773 *
treatment40min/wk 1.8326 1.0247 1.788 0.07371 .
treatment60min/wk 3.6889 1.3134 2.809 0.00497 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 41.589 on 29 degrees of freedom
Residual deviance: 29.458 on 27 degrees of freedom
AIC: 35.458
Number of Fisher Scoring iterations: 4目的変数のeffectの基準カテゴリーは「no」なので、「no」に対するyesの割合がオッズとなります。Interceptの係数=-1.6094が有意なので、「20min/wk」では、「no」に対するyesの確率が、$e^-1.6094=約0.2倍$となり、効果がないことが推定されます。一方、40min/wkおよび60min/wkは、「20min/wk」と比較してオッズが何倍になったのかが推定できます。例えば、60min/wkの係数は$e^3.6889=約40$なので、「20min/wk」に比べるとオッズが40倍になることが推定されます。
結論
20min/wkでは「yes」になる確率が低く、効果が少ないことが示されていますが、治療時間が長くなるにつれて「yes」のオッズが顕著に増加することが示されています。特に60min/wkの効果は非常に顕著で、治療効果が最も強いことが示唆されています。この結果は、より長い治療時間が効果的である可能性を支持しています。
<参考>