

データの準備と要約
必要なパッケージをダウンロードしておきます(ダウンロード方法について)
library(survival)
library(survminer)サンプルを準備します(Rにcsvファイルを読み込む)
dat <- read.csv("surv.csv", header=T, fileEncoding = "Shift-JIS")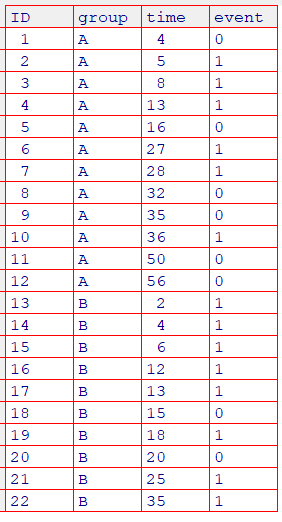
group列: A群=12名、B群=10名
time列: 時期
event列: イベント発生(1: 死亡, 0: 脱落、打ち切り)
カプランマイヤー曲線(Kaplan-Meier曲線)
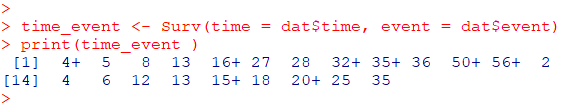
時期とイベントのデータセット
time_event <- Surv(time = dat$time, event = dat$event)
print(time_event )数値は時期、+は「打ち切り」あり
fit <- survfit(time_event ~ group, data=dat)
summary(fit)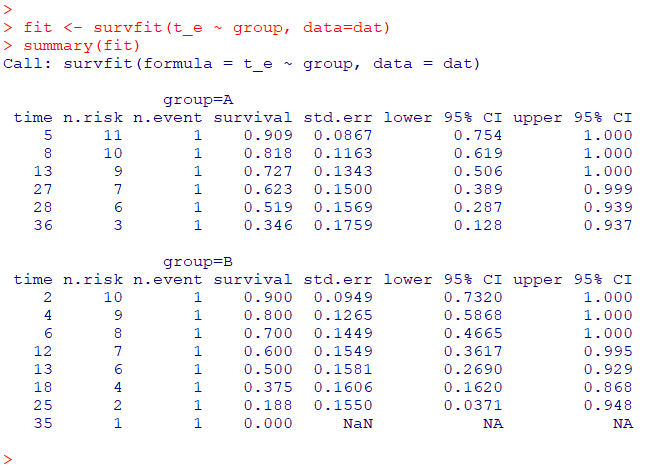
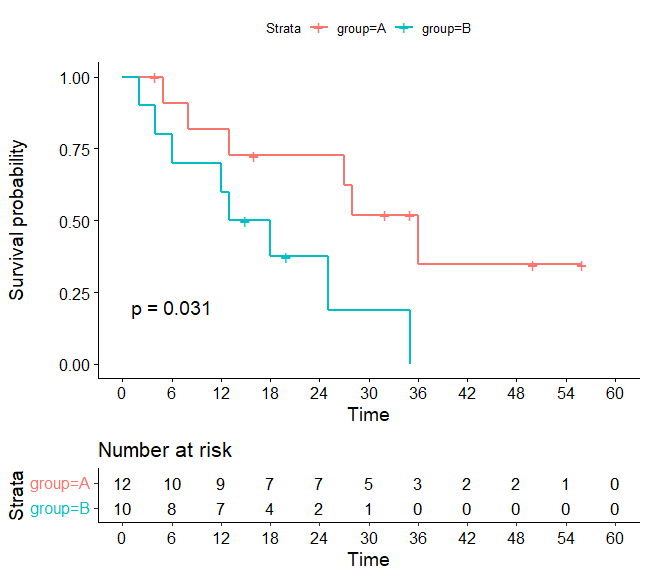
# カプランマイヤー曲線
g1 <- ggsurvplot(
fit, data = dat, pval = TRUE,
risk.table = TRUE, #各時期のリスク集合の数
break.time.by = 6) #x軸の間隔
print(g1)p値はログランク検定の結果
Rでログランク検定(log-rank test)
# ログランク検定の実行
logrank <- survdiff(time_event ~ group, data = dat)
# 検定結果の表示
print(logrank)

p値=0.03となり、有意差が認められました。検定の結果のみでよろしければ、ここまででOKです。ここからは、もう少し詳しく見ていきます。
ログランク検定の検定統計量
時間でソートしたデータ
dat_sorted <- dat[order(dat$time), ]
#View(dat_sorted )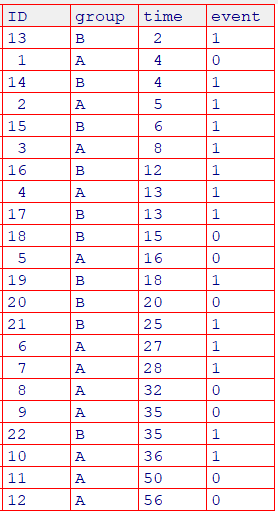
(event; 1=死亡、0=脱落、打ち切り)
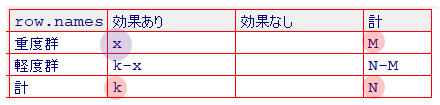
超幾何分布より期待値と分散を求めます
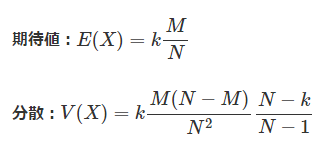
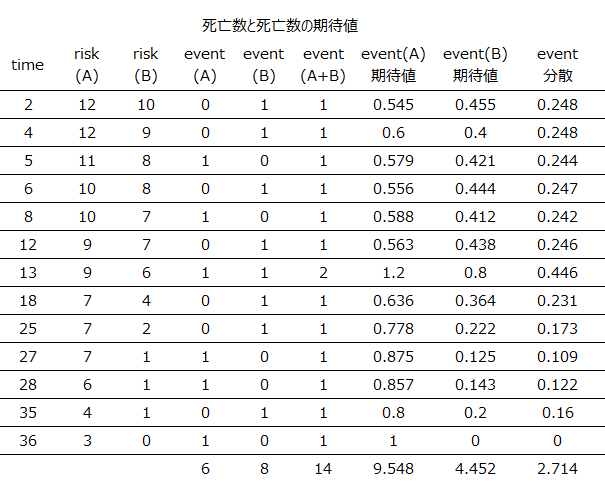
期待値と分散の一覧は以下のようになります(event: 1=死亡、0=脱落、打ち切り)
この一覧を使用して、各検定の統計量が計算されます
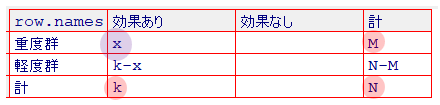
M = risk(A)
N = risk(A) + risk(B)
X = event(A)
k = event(A+B)
Mantel-Haenszel統計量
一般的にログランク検定し使用されている統計量
ログランク検定の検定統計量は、比較されるグループが2つの場合、自由度1のカイ二乗分布に従います
イベント(ここでは「死亡」)が発生する時点で2×2の分割表を作成し、カイ二乗統計量からP値を算出します
各時間での死亡数、期待値、分散から有意な差があるかどうかを判断します
logrank <- survdiff(Surv(time, event) ~ group, data = dat)
print(logrank)
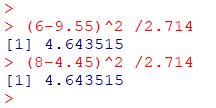
Mantel-Haenszel統計量 = 4.64(自由度1のカイ二乗分布に従う)
期待値と分散の一覧表よりMantel-Haenszel統計量を求める方法
$w_j=1$で重み付けしたカイ二乗統計量
$\chi^2(Mantel-Haenszel) = \dfrac{(\sum(d_{1j}-e_{1j}))^2}{\sum{v_i}}$
(6-9.55)^2 /2.714
(8-4.45)^2 /2.714(A群もB群も同じ解になります)
ログランク検定の統計量
Mantel-Haenszel統計量とは異なる方法でカイ二乗統計量を算出する方法

ログランク統計量($\chi_{LR}^2$)=1.32 + 2.83 = 4.15(自由度1のカイ二乗分布に従う)
期待値と分散の一覧表よりログランク統計量を求める方法
$d_{Ai}$: group A の死亡数
$e_{Ai}$: group A の死亡数を確率変数とした場合の期待値(超幾何分布)
$d_{Bj}$: group B の死亡数
$e_{Bj}$: group B の死亡数を確率変数とした場合の期待値(超幾何分布)
$\chi_{LR}^2=\dfrac{(\sum(d_{Ai}) – \sum(e_{Ai}))^2}{\sum(e_{Ai})}+\dfrac{(\sum(d_{Bj}) – \sum(e_{Bj}))^2}{\sum(e_{Bj})}$
(6-9.548)^2/9.548 + (8-4.452)^2/4.452
参考)重み付けした統計量
混乱しますが、以下のような統計量もあります(他にもあります)
$w_k$で重み付けしたカイ二乗統計量
$\chi^2_{w_k} = \dfrac{(\sum{w_k}(d_{1k}-e_{1k}))^2}{\sum{w_k^2}v_k}$
$d_{1k} : $ イベント数
$e_{1k} : $ イベント期待値
$v_k : $ イベント分散
Rを使用したp値の求め方(参考ページ)
#ログランク検定(method = "1")
surv_pvalue(fit, data = dat, method = "survdiff")
#Gehan-Breslow
surv_pvalue(fit, data = dat, method = "n")
#Tarone-Ware
surv_pvalue(fit, data = dat, method = "sqrtN")method =
“1“または”survdiff” : 通常のログランク検定(Mantel-Haenszel統計量 )、遅い時期の差異を検出するのに敏感
“n“:Gehan-Breslow(一般化ウィルコクソン), $w_k=N$ で重み付け, 早期の差異を検出
“sqrtN“:Tarone-Ware , $w_k=\sqrt{N}$ で重み付け, 早期の差異を検出

詳細については下記論文をご参照ください
- Gehan A. A Generalized Wilcoxon Test for Comparing Arbitrarily Singly-Censored Samples. Biometrika 1965 Jun. 52(1/2):203-23.
- Tarone RE, Ware J 1977 On Distribution-Free Tests for Equality of Survival Distributions. Biometrika;64(1):156-60.
- Peto R, Peto J 1972 Asymptotically Efficient Rank Invariant Test Procedures. J Royal Statistical Society 135(2):186-207.
- Fleming TR, Harrington DP, O’Sullivan M 1987 Supremum Versions of the Log-Rank and Generalized Wilcoxon Statistics. J American Statistical Association 82(397):312-20.



