カプランマイヤー曲線

リハビリテーション研究ではあまり使用されていませんが、カプランマイヤー曲線の基本部分を説明します。カプランマイヤー曲線は、イベントが発生するまでの時間に興味がある場合に利用します。生存率曲線の一種であり、患者が研究から脱落したり、観察期間が終了したりした場合のデータの扱い(打ち切りデータ)を考慮できるため、生存時間データの分析に特化した方法と言えます。
データの準備と要約
使用するパッケージ(パッケージのインストール方法)
.libPaths("C:\\Users\\rehay\\Documents\\Rpackages")
library(survival)
library(survminer)
データはパッケージ survival の lung を使用します
dat <- lung
head(dat)
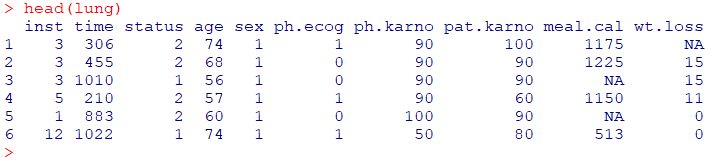
survivalパッケージのlungデータセットは、肺がん患者の生存時間と治療結果を記録したデータです。このデータセットは、肺がんの患者に関する臨床試験のデータを含んでおり、生存分析やコックス比例ハザードモデルなどの統計的手法を用いた研究によく使用されます。具体的には、
lungデータセットには以下のような変数が含まれています:
inst: 患者が登録された研究所の番号time: 生存時間またはフォローアップ時間(日数)status: 生存状態の指標(1 = 死亡、2 = 生存)age: 患者の年齢sex: 患者の性別(1 = 男性、2 = 女性)ph.ecog: ECOGパフォーマンススコア(Eastern Cooperative Oncology Groupのパフォーマンスステータス)。一般的に、スコアが高いほど患者の日常生活活動のレベルが低いことを示します。ph.karno: Karnofskyパフォーマンススコア(医師による患者の活動レベルと能力を評価するスコア)pat.karno: 患者自身によるKarnofskyスコアの評価meal.cal: 一日の摂取カロリー量wt.loss: 最初の診断からの体重減少量(kg)
lungデータセットにおける打ち切りはstatus変数によって表されています。この変数が1の場合はイベント(この場合は死亡)が発生したことを意味し、2の場合は打ち切りを意味します。つまり、statusが2である患者は、研究終了時点で生存していたか、何らかの理由でフォローアップが終了した患者を指します。このデータセットを使用して、様々な生存分析を行うことができます。例えば、生存時間に影響を与える可能性のある要因を特定したり、異なる治療方法の効果を比較したりすることができます。
ChatGPT

打ち切り (censoring):特定のイベント(例えば、患者の死亡や病気の再発)の発生が観測されない状況で研究が終了する場合。研究期間の終了、参加者の研究からの離脱、あるいは別のイベントによる観測の中断などが理由となります。
カプランマイヤー法による生存率曲線(カプランマイヤー曲線)の描き方
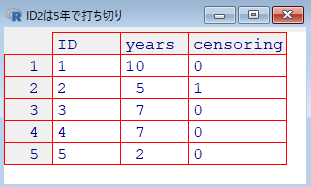
例)5名の生存時間データ
# ID: 患者ID
# years: フォローした年数
# censoring: 生存状態の指標(0 = 死亡、1 = 生存)
ID <- c(1, 2, 3, 4, 5)
years <- c(10, 5, 7, 7, 2)#フォローアップした年数
censoring <- c(0, 1, 0, 0, 0)#生存状態の指標(0 = 死亡、1 = 生存 )
data <- data.frame(ID, years, censoring)
# View(data, title="ID2は5年で打ち切り")
# 生存時間オブジェクトを作成
#event = 1 は、イベントが発生したという意味、つまり死亡ということになります
surv_obj <- Surv(time = years, event = 1 - data$censoring)
#View(surv_obj)
ID2は5年で打ち切り

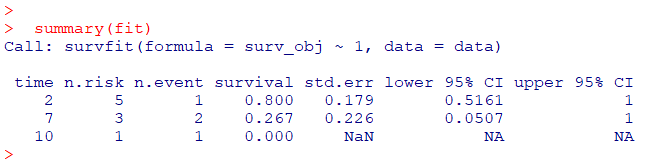
# 層別せずに全体の生存曲線を推定(~1)
fit <- survfit(surv_obj ~ 1, data = data) # data引数を追加
summary(fit)
$N(t)$: 時点tのリスク集合の人数(n.risk)
(リスク集合、リスク集団:時点tにおいてイベントが発生する可能性のある患者の集合)
$d(t)$: 時点tのイベント数(n.event)
ハザード(離散):$\dfrac{d(t)}{N(t)}$
生存関数:$S(t)=s(t_-)×(1-\dfrac{d(t_+)}{N(t_+)}) $ (survival)
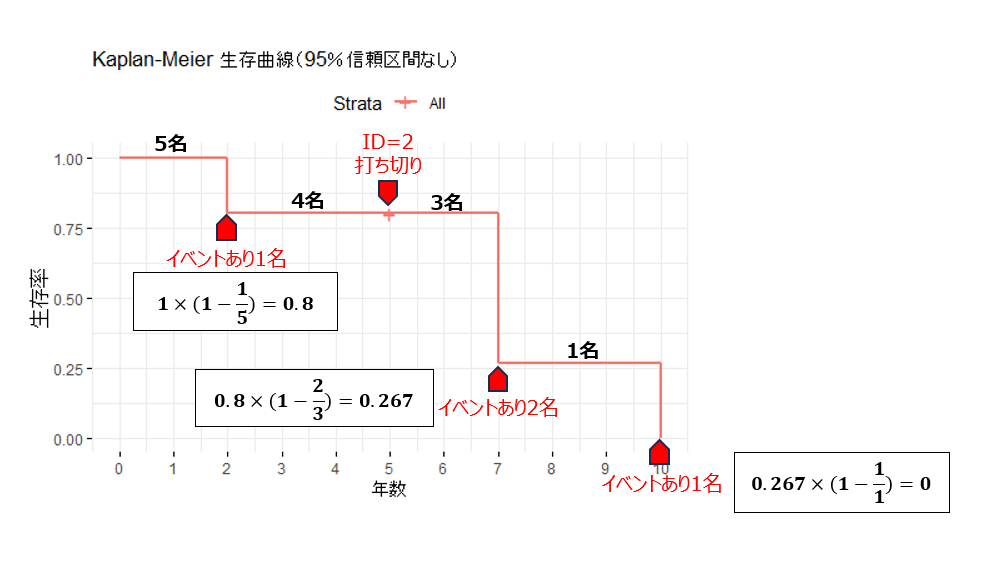
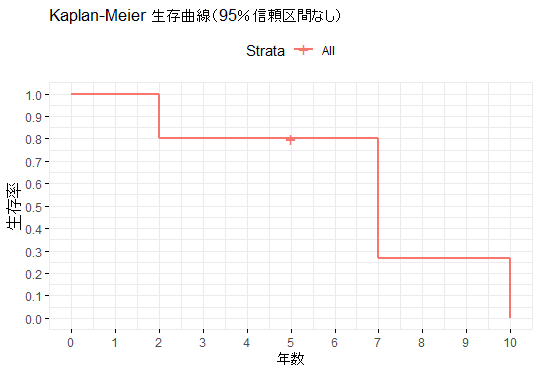
# 生存曲線を描く
g1 <- ggsurvplot(
fit, data = data, conf.int = FALSE,
ggtheme = theme_minimal(), #グラフのテーマ
xlab = "年数", ylab = "生存率",
title = "Kaplan-Meier 生存曲線(95%信頼区間なし)",
break.time.by = 1, break.y.by = 0.1
)
print(g1)
データセット lung より
性別(1 = 男性、2 = 女性)で層別したKaplan-Meier曲線
# 生存時間オブジェクトを作成
surv_obj2 <- Surv(time = dat$time, event = dat$status)
# Kaplan-Meier生存曲線をフィットさせる
fit <- survfit(surv_obj2 ~ sex, data = dat) # data引数を追加
# 生存曲線を描く
g2 <- ggsurvplot(
fit, data = dat,
palette = c("#E7B800", "#2E9FDF"),
ggtheme = theme_minimal(), #グラフのテーマ
pval = TRUE, #log-rank test
conf.int = FALSE, #95%信頼区間の表示
xlab = "日数",
ylab = "生存確率",
title = "性別で層別したKaplan-Meier曲線")
print(g2)
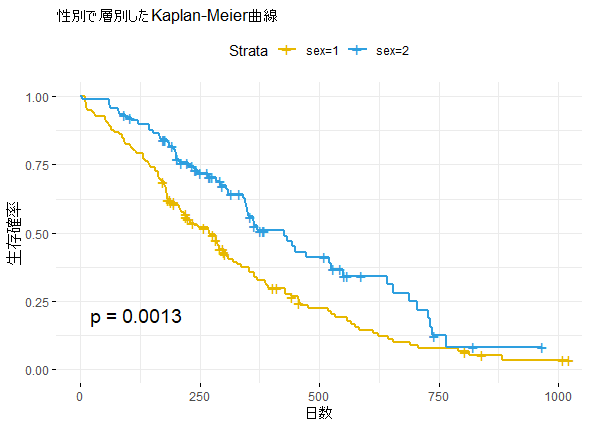
(p値はlog-rank test)
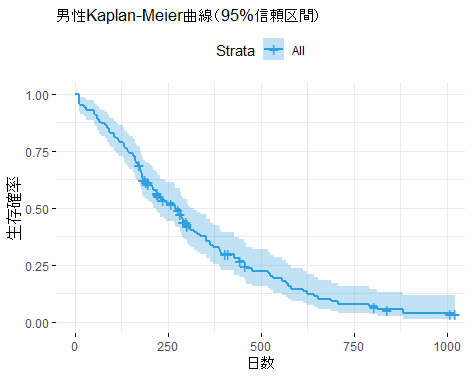
男性のみの曲線と95%信頼区間をプロット
# 男性のデータのみを選択
dat_man <- dat[dat$sex == 1, ]
# 生存時間オブジェクトを作成
surv_obj_man <- Surv(time = dat_man$time, event = dat_man$status)
# 生存分析を実行
km_fit_man <- survfit(surv_obj_man ~ 1)
# Kaplan-Meier曲線を描画
g3 <- ggsurvplot(
km_fit_man,
data = dat_man,
palette = "#2E9FDF", # 男性の色を指定
ggtheme = theme_minimal(),
xlab = "日数",
ylab = "生存確率",
title = "男性Kaplan-Meier曲線(95%信頼区間)")
print(g3)
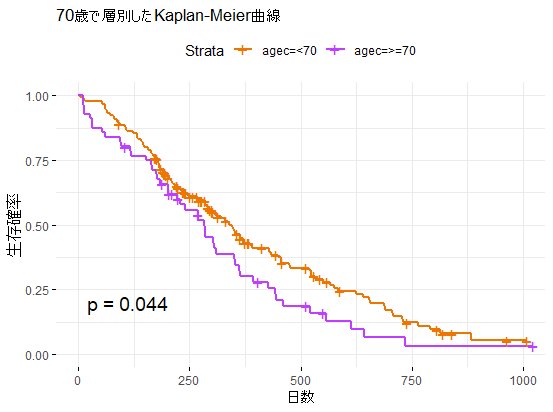
70歳未満と70歳以上の2群をKaplan-Meier曲線で比較
# 生存時間オブジェクトを作成
surv_obj70 <- Surv(time = dat$time, event = dat$status)
# 変数agec (70歳未満を"<70"、70歳以上を">=70")を作成
dat$agec <- ifelse(dat$age < 70, "<70", ">=70")
# Kaplan-Meier生存曲線をフィットさせる
fit70 <- survfit(surv_obj70 ~ agec, data = dat) # data引数を追加
# 生存曲線を描く
g4 <- ggsurvplot(
fit70 , data = dat,
palette = c("#EE7600", "#BF3EFF" ),
ggtheme = theme_minimal(), #グラフのテーマ
pval = TRUE, #log-rank test
conf.int = FALSE, #95%信頼区間の表示
xlab = "日数",
ylab = "生存確率",
title = "70歳で層別したKaplan-Meier曲線")
print(g4)